

| _ID | Prediction | OTHER | SP | mTP | CS_Position | |
|---|---|---|---|---|---|---|
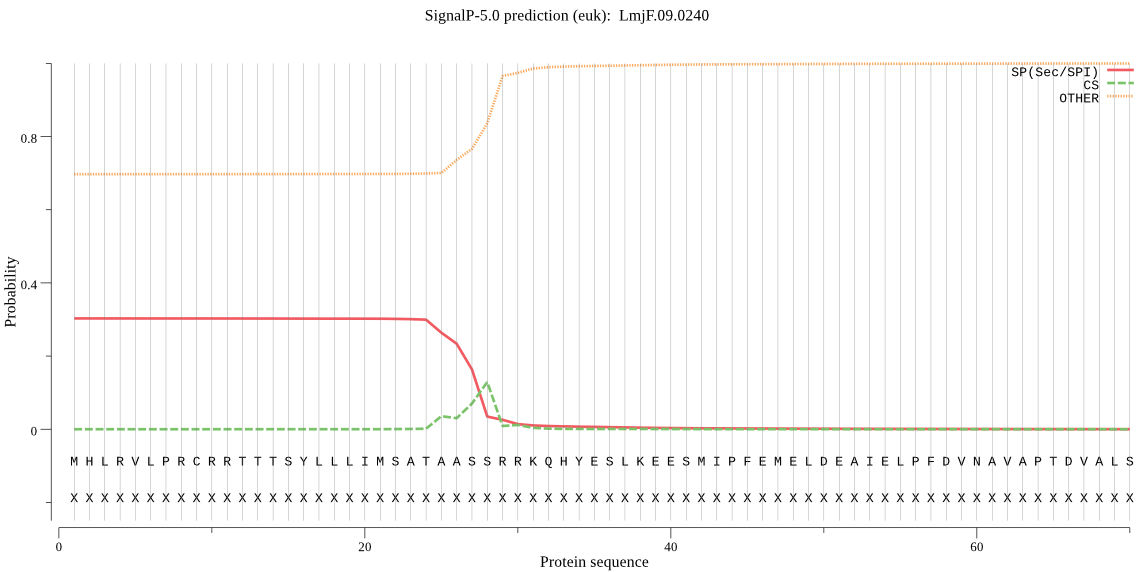
| LmjF.09.0240 | SP | 0.429262 | 0.558285 | 0.012453 | CS pos: 28-29. ASS-RR. Pr: 0.3041 |
MHLRVLPRCRRTTTSYLLLIMSATAASSRRKQHYESLKEESMIPFEMELDEAIELPFDVN AVAPTDVALSAPFGGKKIPMRAARAASESRVAWSNGSTASVGFAVCGNGKELPSTAATAD GVASPIATPANAAPTTYSAAAPSRMLTWKDTSASQRRAEAQASRQDDFHPMVLCPLQNLG NTCYFNAGVQLLVNCPALVYAVRNSPFARWTRSNGPLTAQAVPMPRAEAAPAATKLCLKG GAAIHTLFQEFSILLARMEMGCAAGERAISPICALDSLAQAYPQFEGRSQQDAAEMMTSL LASLEEEGGQYVEVAQLLQSFEDAAALRQGVTPLASSPRASDPVTRALKGATSPLLEAET APSASPHRRNYSEPCNIEEYSQSSLLSSTSEDTFLPGPRSAHFFNVLRLMERVNRENELL ERRVKQRQGIAYTNSFKPPRLHFNPLLDGFRGYTLSEVECHNCRAVSRVVSPFNGLLLDV PTAKQRRRYALAHPNVPRRVDFDGQLRQVKKPFRLTWWNPLSLCVAAWRQLKQFFQDPFP YPLWLDECLDIHFEPEVLRGCSRYRCESCDTTTDATKSETLLALPEYLLIYMKRFEAGRF FNSKKSDSVFFPTSWRPLTTTQARQSPINTDDAPPALPEYLDLRHYLHSSVAPFAEPIPP CLWGTDNEPHQQHHVSRDDASTPTSSIATTYTLDGIVNHHGGYDGGHYTVFLYKATEERQ AWVYISDDEVEQAEEVVATDTEYVLLYRRQPLVQAAPESDEAEQLRRKACYYLSQSSPSC PAPNAAAATTDGPKASKPHSSCPFSSSSAGGVASTPPHAATAATEPWVYISRMWLQRVAF MHEPGPIVNRLCYCRPEDRGRVSMYQSIFPATVKVPAEVPHVHGPPVSWFYAPITQRDYD IFYNAFGGNLAVTASEYESLRAMQDKFCAAIDLAERQRGSAARPAPRH
| ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | ID | Site | Peptide | Score | Method | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LmjF.09.0240 | 154 S | DTSASQRRA | 0.996 | unsp | LmjF.09.0240 | 154 S | DTSASQRRA | 0.996 | unsp | LmjF.09.0240 | 154 S | DTSASQRRA | 0.996 | unsp | LmjF.09.0240 | 270 S | ERAISPICA | 0.995 | unsp | LmjF.09.0240 | 303 S | SLLASLEEE | 0.995 | unsp | LmjF.09.0240 | 320 S | QLLQSFEDA | 0.993 | unsp | LmjF.09.0240 | 337 S | PLASSPRAS | 0.997 | unsp | LmjF.09.0240 | 341 S | SPRASDPVT | 0.992 | unsp | LmjF.09.0240 | 388 S | SLLSSTSED | 0.99 | unsp | LmjF.09.0240 | 626 S | QARQSPINT | 0.998 | unsp | LmjF.09.0240 | 863 S | RGRVSMYQS | 0.992 | unsp | LmjF.09.0240 | 36 S | QHYESLKEE | 0.997 | unsp | LmjF.09.0240 | 147 T | SRMLTWKDT | 0.99 | unsp |